
本次分享的文章是Asim Iqbal等人2019年发表在Nature Machine Intelligence上的文章《Developing a brain atlas through deep learning》.
神经科学家已经投入了大量的努力来创建标准的大脑参考图谱,从而来高通量配准感兴趣的脑解剖区域。题目中所讲的图谱绘制是指利用配准的方式,将小鼠的脑配准到标准模板上。针对这一目的,所需要面对的挑战主要来自于个体之间脑的大小和形状存在差异。
该工作的贡献可概括为以下四方面:
- 以较少的人工干预(标注)对脑的感兴趣区域(ROI)进行分割;
- 利用文章提出的pipeline绘制老鼠不同发育阶段的图谱;
- 探究了一系列的神经标记和成像模态;
- 将算法利用到人脑的磁共振影像中,探究其性能。
传统high-throughput image registration frameworks存在以下几个问题:
- 大多方法都依赖于人工干预(semi-manual;参数的设定,比如强度阈值、背景对比度等);
- 将脑切片配准到参考图谱上(reference atlas)的能力有限。
要利用深度学习实现文章的目标,可分解为切片中脑区域的识别(分类任务)以及识别后与标准模板的配准。在数据量方面面临困难在于需要数据量较大的、参照标准模板中标签(脑区)进行标注的脑切片数据集。
如之前所提到的,这项工作的目的在于将脑切片配准到标准模板,从而能够识别出脑切片中某一位置隶属于标准模板中的哪一个区域,因此,该目标可以转化为对脑切片进行分割(Segmentation)任务,从而实现确定切片上每一个像素点属于标准模板上的哪一类。整体pipeline如图1所示。

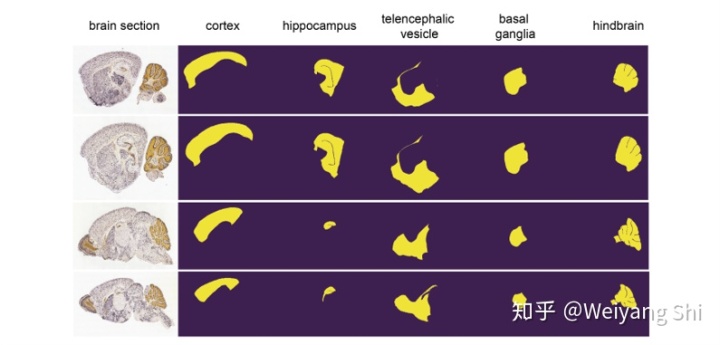
文章采用了在COCO数据集上预训练的Mask R-CNN模型来对不同发育时间段内的鼠脑(以及人脑)进行segment和annotate。最后通过模型输出结果与真实标注、传统的配准或分割方法进行对比来评估模型性能,如图2所示。
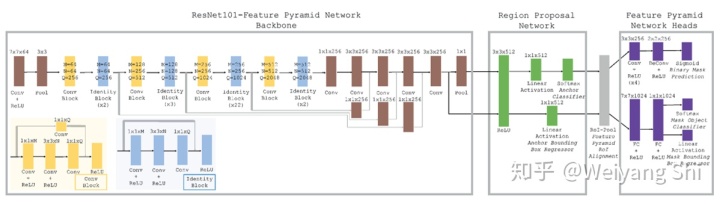
从图一不难看出,模型最后优化的是一个多任务的损失函数:
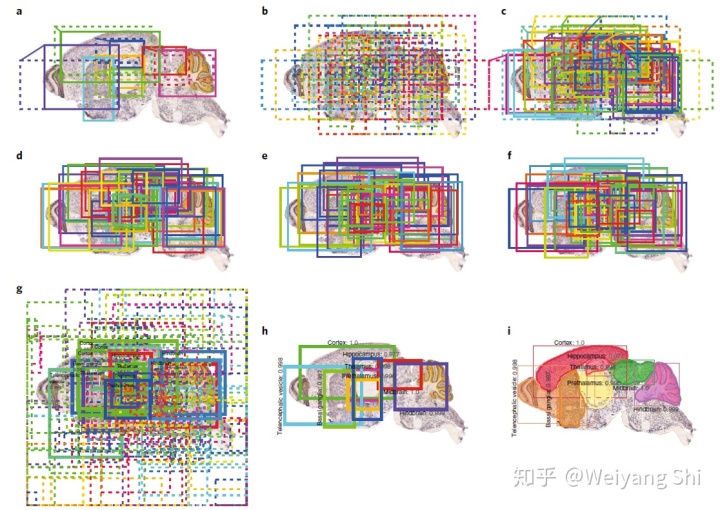
如图4所示,这一框架的图像处理流程可以由a-i展示,其中a-f代表大脑区域定位过程,g-h代表对提出的锚框进行分类,i则代表最后的分割(binary mask).
文中总结指出这种方法标志着处理大规模脑数据集的范式转变,它可以处理任意层的切片,而独立与人工选择干预。
事实上,深度学习确实提高了生物医学领域对于大数据(单个数据维度大)的处理能力,越来越多的基础研究开始利用深度学习去代替一些重复性、耗时、耗力但有标签的模式识别问题。但更值得我们探究的问题或许在于:
- 在生物医学领域,样本处理方式在不同实验室、不同仪器中存在一定的差异,因此一个模型是否拥有良好的泛化性能,还是说需要在不同的实验设定下重新标注样本进行训练?由于生物医学领域的特殊性,样本的获取及标注都存在更大的代价,因此对于鲁棒性模型研究以及小样本学习的期望理应更高;
- 发展针对该领域的无监督学习,或许能够为我们提供更多值得验证的发现。