libsvm的安装
libsvm是一个实现了SVM支持向量机的库,可以在MATLAB上配置调用,以下是我的安装流程,包括记录一下在安装过程中遇到的一些问题。我的版本是MATLAB R2019b,在网上能找到下载安装的方法,在此不赘述。建议最好在2017版本及以上,我不确定后面MinGW的安装会不会出现不兼容情况。
进入官网,拉到中间找到下载链接。你可以下载zip格式然后解压到…\MATLAB R2019b\toolbox目录下。
MATLAB转到该目录下,命令行输入>>make即可编译,但一般情况下会报错。如果遇到这个错误:
需要打开环境->附加功能->获取附加功能,搜索MinGW并安装。我第一次安装总是下载不成功,我的解决办法是先点击下载,这时文件是可以成功下载到默认目录的,然后再点击一次安装,跳过前面的下载步骤就快多了。
当然,安装好编译器只能算成功了一半,输入make之后大概率会遇到下面这个报错: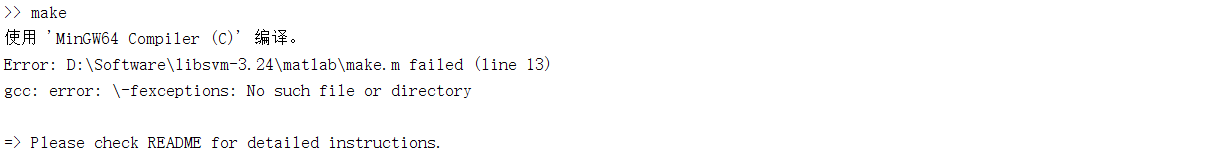
这里推荐一篇文章MATLAB安装配置libsvm(附MinGW64配置),完美解决我的问题。
好了,现在你已经安装好了libsvm,可以开始大展身手了。
官方教程
首先祭出官方教程,SVM原理讲解很详细,目前正在研究中。下面提一下对我最有启发性的一点讨论。
很多初学者的实验步骤是这样的:
- 将实验数据处理成SVM包接受的格式
- 随意选择核函数和参数训练
- 测试
显然这是不严谨的,如果效果好纯粹是瞎猫碰到死耗子。官方给出的建议是:
- 将实验数据处理成SVM包接受的格式
- 对数据进行简单缩放
- 考虑RBF核 K ( x , y ) = e − γ ∣ ∣ x − y ∣ ∣ 2 K(x,y)=e^{-\gamma||x-y||^2}K(x,y)=e−γ∣∣x−y∣∣2
- 通过交叉验证确定最优的 C CC 和 γ \gammaγ
- 使用最优参数训练整个训练集
- 测试
这么做的好处是显而易见的,官方甚至出了张效果对照表,科学地论证了一个有理有据的参数选择多么重要。
| 应用 | 训练集 | 测试集 | 属性 | 类别 | 初学者的准确率 | 高手的准确率 |
|---|---|---|---|---|---|---|
| 天体粒子 | 3089 | 4000 | 4 | 2 | 75.2% | 96.9% |
| 生物信息学 | 391 | 0 | 20 | 3 | 36% | 85.2% |
| 交通工具 | 1243 | 41 | 21 | 2 | 4.88% | 87.8% |
从这张表还是可以看出来,使用数据缩放和交叉验证之后准确率的提升还是令人非常印象深刻的。
一个简单的测试实例
官方教程以SVM原理为主,想尽快找个代码跑起来的初学者可能会看得一头雾水。下面是我从xiaopihaierletian的博客偷师学来的代码,让我能以最基础的参数设置快速上手。在这里我复述一下,欢迎去原博客支持一下。
首先原博客文中提到要将svm开头的文件改名成libsvm开头,防止重名。我这里改过后反而报错,想来是文件中的调用没有彻底改掉,我就没有改名了,所以下面的代码和原博客稍微有点不同。读取官方自带的heart_scale数据,只能用libsvmread而非load,原因是这是C++格式的数据,后者会导致报错。当然你也可以下载MATLAB格式的数据,我们这里就不麻烦了,换个函数就好。进入LIBSVM的根目录运行以下代码:
[heart_scale_label, heart_scale_inst] = libsvmread('heart_scale');
model = svmtrain(heart_scale_label, heart_scale_inst, '-c 1 -g 0.07');
[predict_label, accuracy, dec_values] = libsvmpredict(heart_scale_label, heart_scale_inst, model);
第二行代码敲进去之后界面会出现这样的反馈:
第三行代码跑出来这样的结果:
结果出来了,好开心!现在你已经翅膀硬了,可以在SVM的世界自由探索了。