过去十年来,由于DNA甲基化芯片技术的不断发展以及测序成本的快速下降,DNA甲基化芯片数据呈现爆发式增长。这些数据是表观基因组关联研究(Epigenome-Wide Association Studies,EWAS,NAR| 表观组关联分析数据库 - EWAS Data Hub)的宝贵资源,为基于大规模整合分析的EWAS研究提供了数据支撑。然而,在整合公共DNA甲基化芯片数据时,不得不面对两个棘手的问题。首先,公共数据样本量大且增长速度快,必须要考虑大数据整合中的N+1问题。其次,大多数公共数据没有提供原始数据。因此,无法获取control探针和OOB (Out Of Band)探针的信息,而这两类探针的信息是目前绝大多数DNA甲基化芯片标准化方法必须的。
为此,北京基因组研究所(国家生物信息中心)国家基因组科学数据中心开发了一种基于参考的高斯混合模型分位数标准化方法 (Gaussian Mixture Quantile Normalization, GMQN),该方法以被广泛使用的芯片内标准化方法SWAN和BMIQ为基础进一步对DNA甲基化芯片数据进行标准化,特别是去除批次效应。该方法以“GMQN: A Reference-Based Method for Correcting Batch Effects and Probe Bias in HumanMethylation BeadChip”为题于2022年1月在frontiers in genetics期刊上在线发表。

为了去除批次效应等偏差,我们首先需要找到这些偏差在数据中的表现形式。为此,我们对不同类型探针的信号值分布特征进行了探索。我们发现一类探针红绿信道的信号值都可以分解为两个高斯分布的叠加(图一 a, b, c, d),且这些高斯分布的拟合参数能够很好地区分批次(图一e)。利用这一特性,我们借鉴BMIQ的思想,对一类探针红绿信道的信号值分别拟合高斯混合分布,再将不同样本对应高斯分布的形状调整至相同以此降低批次效应和其他偏差。
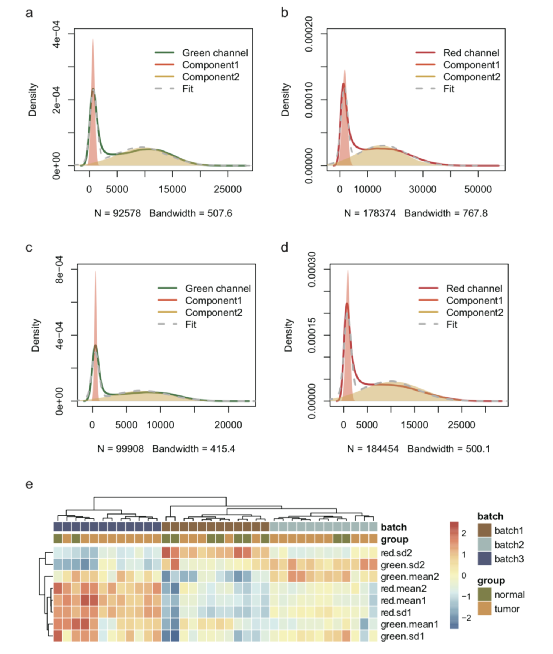 图一、Infinium I探针的信号强度分布特征和基于高斯分布拟合参数的不同批次样本的聚类结果
图一、Infinium I探针的信号强度分布特征和基于高斯分布拟合参数的不同批次样本的聚类结果
为了检验GMQN是否能否进一步提升SWAN和BMIQ的标准化效果,我们设计了四种基准测试(图二),即大量技术重复样本的批次效应的检测(图二a, b)、两种大样本疾病case/control研究(图二c, d)、以年龄性状为代表的回归分析(图二e)和临近位点甲基化水平的比较(图二f)。通过以上四种不同应用场景下进行测试,我们发现GMQN能够有效降低公共数据中的噪音,提高下游分析的准确度。
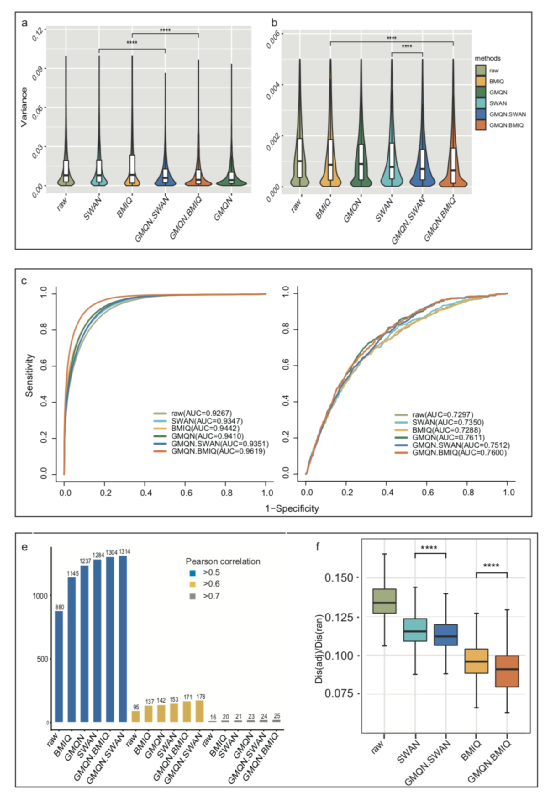 图二、GMQN的基准测试结果
图二、GMQN的基准测试结果
目前,EWAS Open Platform(https://ngdc.cncb.ac.cn/ewas/)中收录的115,852个样本以及MethBank(https://ngdc.cncb.ac.cn/methbank/)中的参比甲基化图谱构建的数据均采用该方法进行整合和标准化。
为了统一和对接绝大多数分析流程,GMQN采用R语言编写,并构建了gmqn R包。用户可以通过devtools::install_github('MengweiLi-project/gmqn')安装并使用。
下面展示GMQN的使用流程(输入文件格式见图三):
1、如果有idat原始文件, 我们建议使用minfi包来读取,得到m和um文件。
library(minfi) #不存在该R包,需安装
RGSet = read.metharray.exp("idat/") #“idat”文件下存放的是所有的idat文件
MSet <- preprocessRaw(RGSet)
m = data.frame(getMeth(MSet))
um = data.frame(getUnmeth(MSet))2、如果没有原始文件,可以直接输m和um文件,可以使用gmqn来标准化得到beta值文件。
library(gmqn)
ref = set_reference(m, um) #如果使用默认的ref,跳过该行执行
###可以选择结合swan或bmiq方法的标准化,ncpu可以指定线程数
beta.GMQN.swan = gmqn_swan_parallel(m, um, ncpu = 4, ref = ref)
beta.GMQN.bmiq = gmqn_bmiq_parallel(m, um, ncpu = 4, ref = ref)GMQN具体使用方法和详细代码见:https://github.com/MengweiLi-project/gmqn.

图三、GMQN输入文件格式示意图
北京基因组研究所(国家生物信息中心)博士研究生熊壮以及博士毕业生李萌伟为共同第一作者,鲍一明研究员为论文通讯作者。该研究得到了中科院战略性先导科技专项、国家重点研发计划、中科院关键技术人才等项目资助。
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集