在基因数据的分析中,经常会用到lasso(套索)这种方法来进行变量的筛选,其实就是在目标优化函数后边加一个L1正则化项,其中参数lamda为超参数,需要我们去确定。接下来以线性回归为例介绍其在R语言中的实现,当然在logistic回归、cox回归也是可用lasso的。
实例数据
data("longley")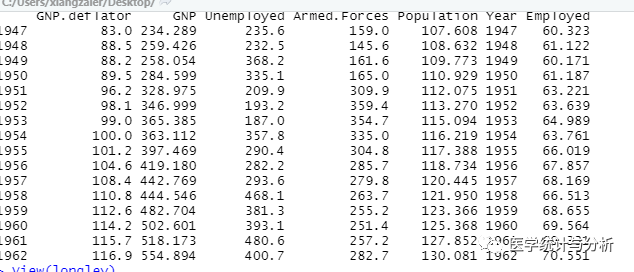
R包(glmnet)
library(glmnet)我们用交叉验证来确定lamda的值,在这里我们随便指定使用nfolds=4 4折交叉验证,实际中常用5折或者10折。以Employed为因变量,其余变量为自变量(不包括年份)建立模型
set.seed(123)x <- as.matrix(longley[,-c(6,7)])cv_lasso = cv.glmnet(x, longley$Employed,nfolds = 4,family = "gaussian", alpha = 1)在这里对应于mse最小的lamda即为我们最终采用的lamda,lambda.min就是所求,大小约为0.097
plot(cv_lasso) sprintf('Best lambda for LASSO: %f.', cv_lasso$lambda.min)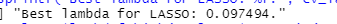
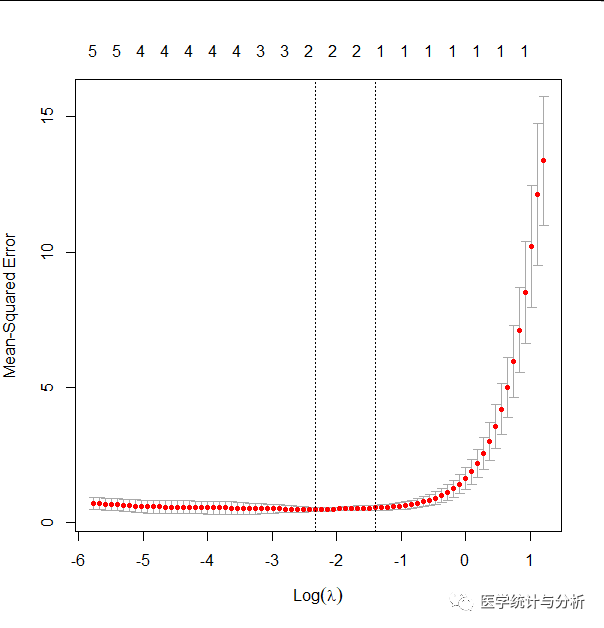
我们可以验证下看lambda.min对应的是不是最小的mse,运行下面程序,发现mse最小处对应的lamda与以上程序返回值一致
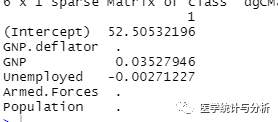
cv_lasso$lambda[which(cv_lasso$cvm==min(cv_lasso$cvm))]返回参数的系数,可以看到只剩下两个变量
coef(cv_lasso,s = "lambda.min")