1.概述
文章主要设计了一种端到端的模型(GNN-PPI),用于预测PPI(蛋白质相互作用)网络的边(作用)类型。
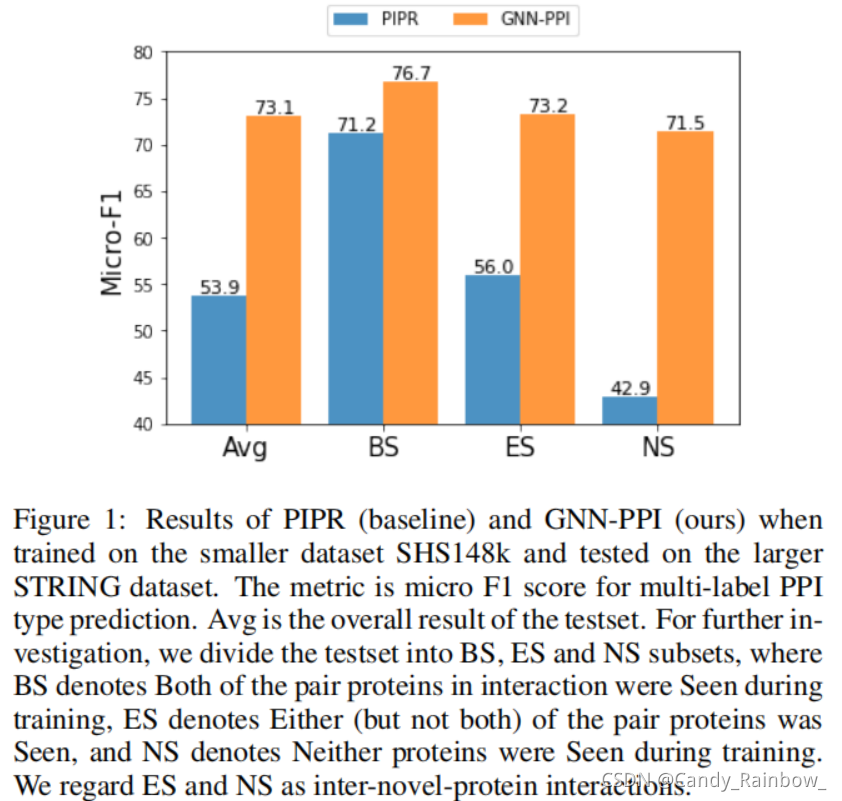
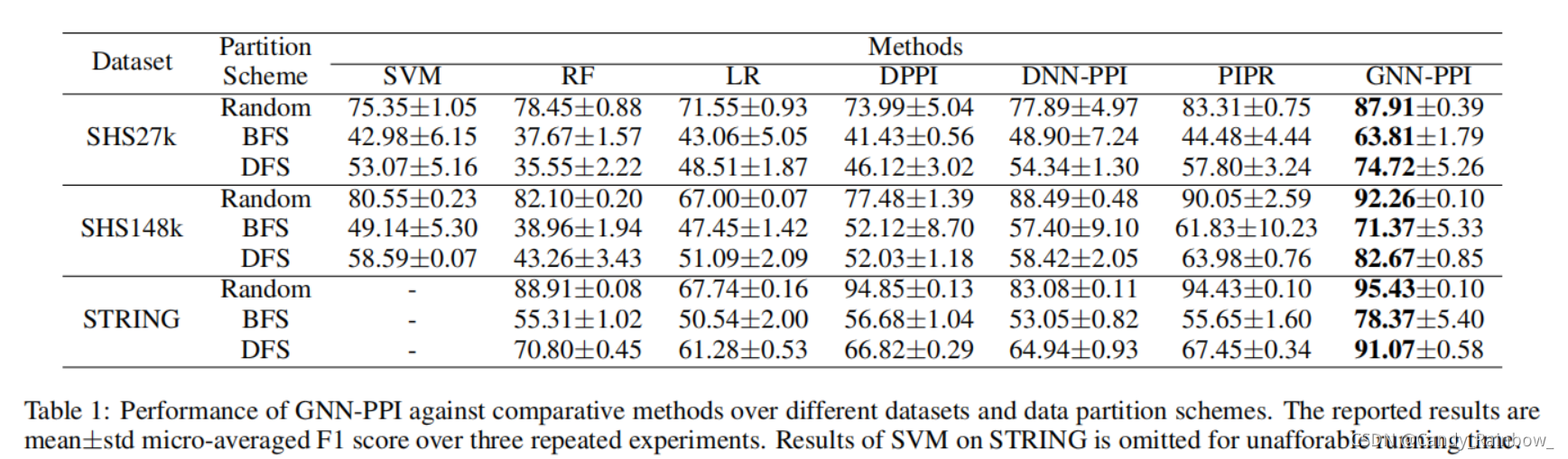
主要与2019年的PIPR做对比,性能提升明显。
2.贡献
1.设计了一个新的评估框架,充分尊重新蛋白之间的相互作用,并在数据集上给出一致的评估。(新蛋白质指的是训练集中没有出现的蛋白质)
2.建议将蛋白质之间的相关性纳入PPI预测问题。提出了一种基于图神经网络的关联建模方法。
3.提出的GNN-PPI模型在不同尺度的真实数据集中达到了最先进的性能,特别是在新蛋白相互作用的预测。
3.方法
3.1问题建模
符号定义
蛋白质集合 ![]()
PPI集合 ![]()
蛋白质标签集合 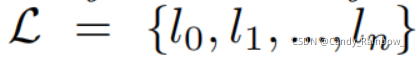
对于任意的标签
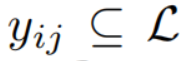
多类型PPI数据集 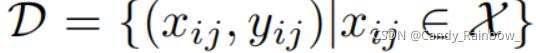
蛋白质作为节点,PPIs作为边构成PPI图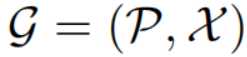
模型任务
多类型PPI学习 : 从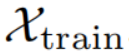 中学习到
中学习到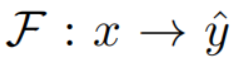
对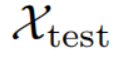 中的任意
中的任意预测出标签(一个或者多个)
3.2数据集划分
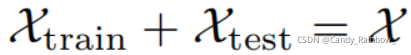
蛋白质集 被划分成:
已知
未知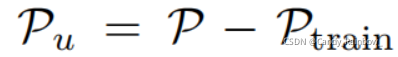
测试集被划分成:
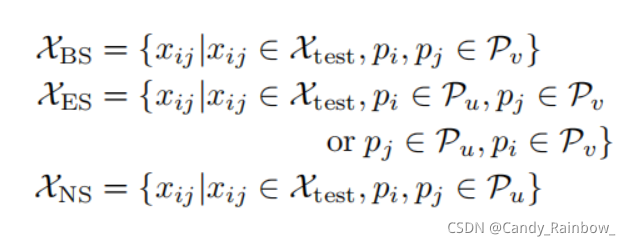
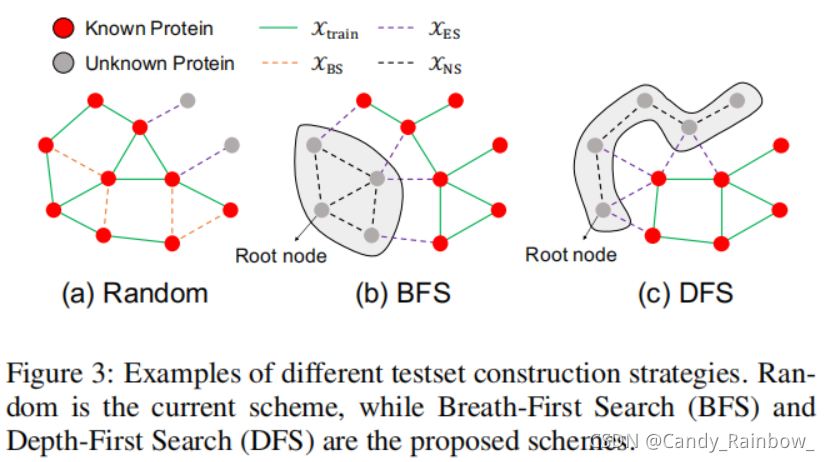
测试集选取策略:
伪代码:

条件: BS << ES + NS
采用随机法,体现不出鲁棒性
3.3模型设计
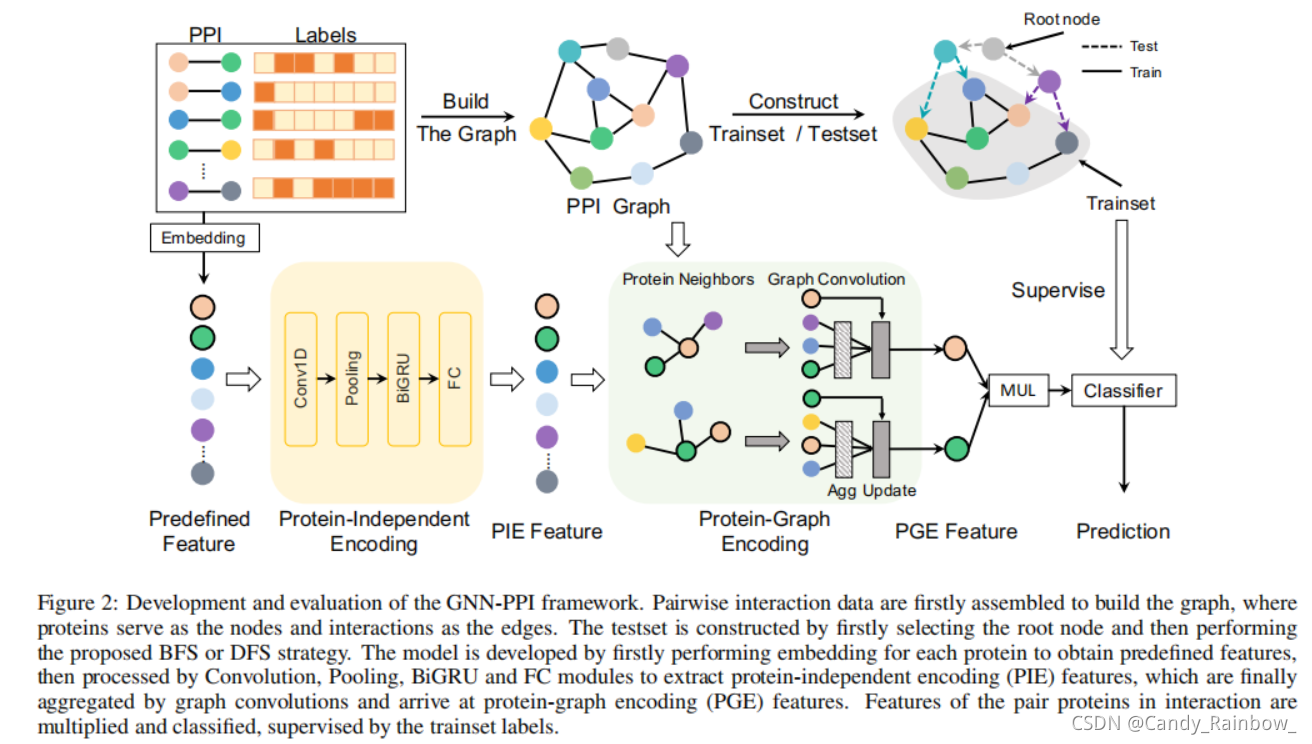
输入:
蛋白质集,边集,标签集
模型拆分:
1.评估板块
拆分训练集,测试集。
2.编码板块
蛋白质特征编码(蛋白质独立编码PIE->蛋白质图编码PGE)。
PIE利用氨基酸序列信息编码,并作为PPI网络的输入,PGE根据PPI网NN结合邻居信息,得到进一步的表征。
将原独立的学习任务 转换成
转换成
 。
。
GNN的第k层:
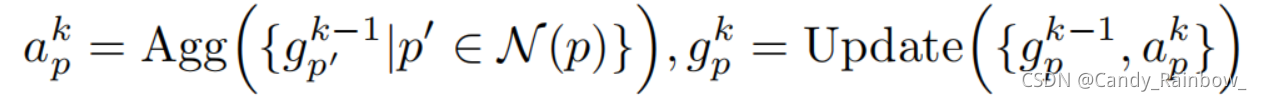
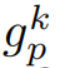 表示节点p在第k层特征
表示节点p在第k层特征
本文采用的是GIN
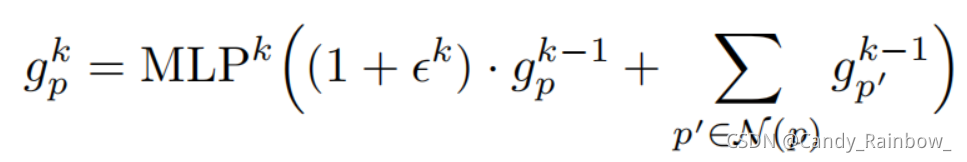
ε是一个可学习的参数或者是固定值
3.预测板块
对于未知的PPI,结合前一过程编码的蛋白质特征,计算它们在不同PPI类型中的得分,并输出其多标签预测。
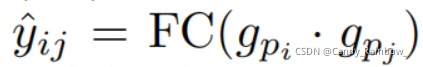
损失函数(二元交叉熵)
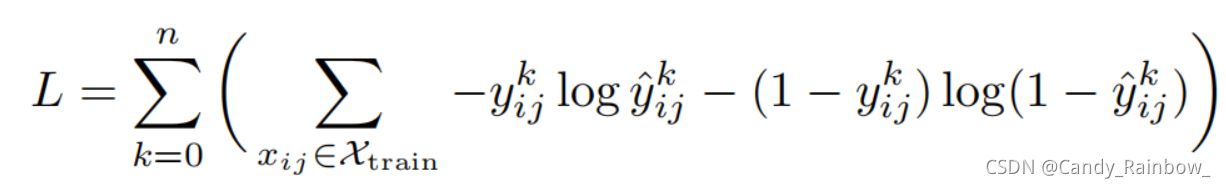
4.实验
版权声明:本文为Candy_Rainbow_原创文章,遵循CC 4.0 BY-SA版权协议,转载请附上原文出处链接和本声明。