随机森林简介
如果读者接触过决策树(Decision Tree)的话,那么会很容易理解什么是随机森林。随机森林就是通过集成学习的思想将多棵树集成的一种算法,它的基本单元是决策树,而它的本质属于机器学习的一大分支——集成学习(Ensemble Learning)方法。随机森林的名称中有两个关键词,一个是“随机”,一个就是“森林”。“森林”我们很好理解,一棵叫做树,那么成百上千棵就可以叫做森林了,这样的比喻还是很贴切的,其实这也是随机森林的主要思想—集成思想的体现。“随机”的含义我们会在下边部分讲到。
其实从直观角度来解释,每棵决策树都是一个分类器(假设现在针对的是分类问题),那么对于一个输入样本,N棵树会有N个分类结果。而随机森林集成了所有的分类投票结果,将投票次数最多的类别指定为最终的输出,这就是一种最简单的 Bagging 思想。
更多关于此方法在宏基因组学中的应用,请阅读之前分享的文章:
R randomForest包
randomForest包主要功能是分类和回归分析,一共提供了39个函数,最常用的就是randomForest来实现分类(Classification)和时间序列回归(Regression)
今天我们先讲最常用的分类方法(用于分组的特征Features),下周再讲解回归的应用(时间序列预测模式,如预测股票、尸体死亡时间等)。
安装与加载# 安装
install.packages("randomForest")
# 加载
library(randomForest)
分类Classification
先了解一下输入数据格式,方便准备
使用R内置鸢尾花数据data(iris)
head(iris)
数据包括150个样品,4列属性数据,1列分组数据。Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
设置随机数种子保证结果可重复set.seed(315)
随机森林分类iris.rf = randomForest(Species ~ ., data=iris, importance=TRUE, proximity=TRUE)
显示结果,默认使用500个树,获得两个变量分离样品,错误评估矩阵print(iris.rf)
结果如下:包括分析的命令,优化选择的分类变量个数2,和数据再分类和错误率统计结果。此例 中使用2个变量分类,三种花的分类错误率为4%,每组中分类结果和错误率详见表格。Call:
randomForest(formula = Species ~ ., data = iris, importance = TRUE, proximity = TRUE)
Type of random forest: classification
Number of trees: 500
No. of variables tried at each split: 2
OOB estimate of error rate: 4%
Confusion matrix:
setosa versicolor virginica class.error
setosa 50 0 0 0.00
versicolor 0 47 3 0.06
virginica 0 3 47 0.06
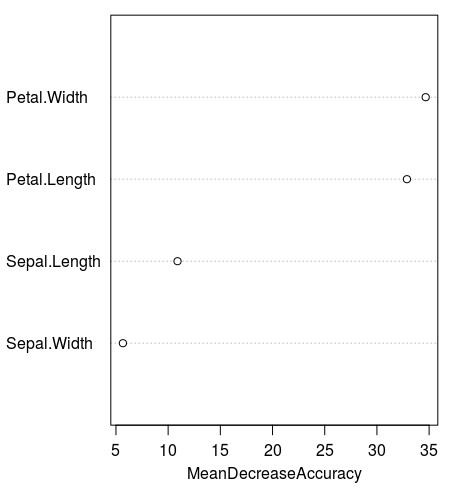
查看每个变量的分类贡献度,显示仅保留两位小数可读性更好round(importance(iris.rf), 2)
setosa versicolor virginica MeanDecreaseAccuracy MeanDecreaseGini
Sepal.Length 5.59 7.50 8.27 10.90 8.80
Sepal.Width 4.88 1.54 5.20 5.67 2.66
Petal.Length 21.03 33.20 27.24 32.87 40.62
Petal.Width 23.46 34.17 32.89 34.67 47.09
可视化MeanDecreaseAccuracy,直译为平均减少准确度,即没有这个Feature时,分类准确度下降的程度,相当于我们常用的分类贡献度的概念。varImpPlot(iris.rf)
分类结果主坐轴分析## 分类结果主坐轴分析和可视化 Do PCoA/MDS on 1 - proximity:
iris.mds= cmdscale(1 - iris.rf$proximity, eig=TRUE)
# 设置显示样品点,而不是变量点
op= par(pty="s")
# 散点图展示每个Feature与PCoA1/2轴间的相关分布
pairs(cbind(iris[,1:4], iris.mds$points), cex=0.6, gap=0,
col=c("red", "green", "blue")[as.numeric(iris$Species)],
main="Iris Data: Predictors and MDS of Proximity Based on RandomForest")
par(op)
print(iris.mds$GOF)
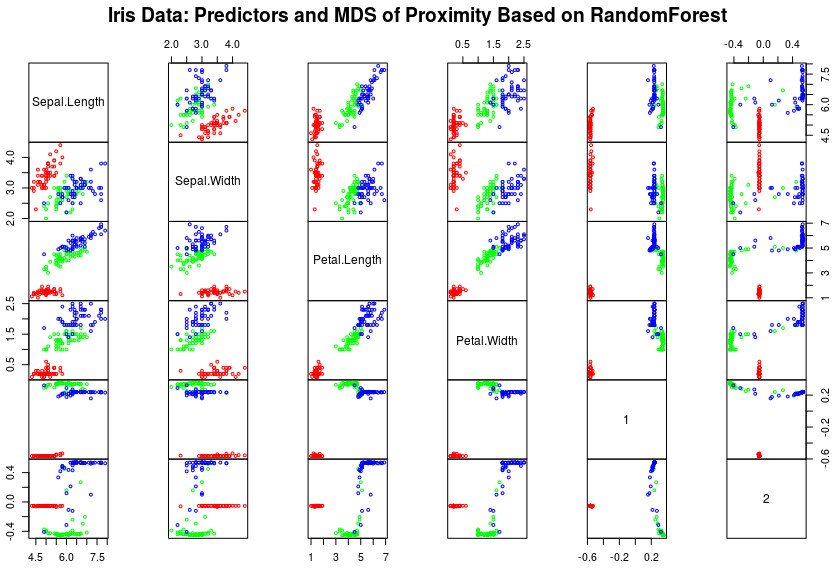
可以看到各Feature间的相关性,如Petal.Width与Petal.Length正相关非常好,而且大小也可以很好分开不同组;更可以看到PCoA轴1/2与每个Features分布样式,是如何区分组的。
随机选取2/3预测,1/3验证# 随机1-2有放回抽样150次,概率分别为0.67和0.33,用于获取训练集
ind=sample(2,nrow(iris),replace=TRUE, prob=c(0.67,0.33))
# 2/3作预测建模
iris.rf = randomForest(Species ~ ., iris[ind==1,], ntree=1000,
nPerm=10, mtry=3, proximity=TRUE, importance=TRUE)
print(iris.rf)
# 1/3验证预测模型
iris.pred = predict(iris.rf, iris[ind==2,] )
# 输出预测与观测对应表
table(observed=iris[ind==2,"Species"], predicted=iris.pred)
我们使用末被用于建模的1/3数据进行验证,更可信。结果如下。predicted
observed setosa versicolor virginica
setosa 18 0 0
versicolor 0 16 1
virginica 0 2 16
无监督分类## 无监督分类 The `unsupervised' case:
set.seed(315)
iris.urf= randomForest(iris[, -5])
# 主坐标轴分析并展示
MDSplot(iris.urf, iris$Species)
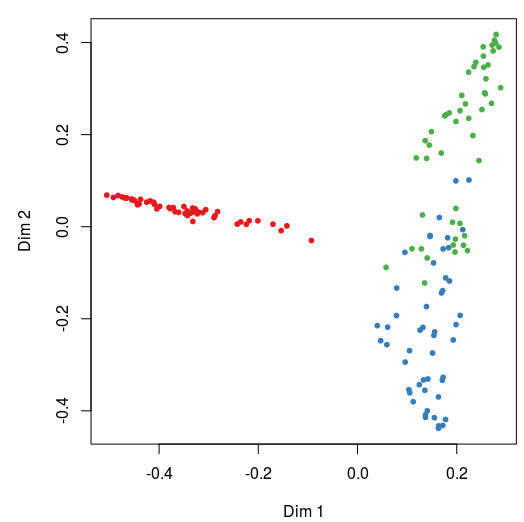
可以看到,其实3个物种在分组末知的情况下,也是可以非常好的分开的。其实在分析前我们应该进行PCoA,查看组间是否存在差异,再进行随机森林找Features。
分层抽样#stratified sampling: draw 20, 30, and 20 of the species to grow each tree.
(iris.rf2= randomForest(iris[1:4], iris$Species, sampsize=c(20, 30, 20)))
结果如下:Call:
randomForest(formula = Species ~ ., data = iris, importance = TRUE, proximity = TRUE)
Type of random forest: classification
Number of trees: 500
No. of variables tried at each split: 2
OOB estimate of error rate: 4.67%
Confusion matrix:
setosa versicolor virginica class.error
setosa 50 0 0 0.00
versicolor 0 47 3 0.06
virginica 0 4 46 0.08
随机森林自动评估,利用袋外数据评估错误率也是非常可靠的(OOB),所以可以不人为分组进行验证。
但严谨总没有坏处,好的结果都是多角度证明的。
大家学习随机森林的分类、和下周要讲的回归。将带大家上手重复一些高水平文章中的分析,结合具体生物学问题会更有意思。
Reference
猜你喜欢
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外1500+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。

学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
