基因表达谱热图的绘制
所需数据格式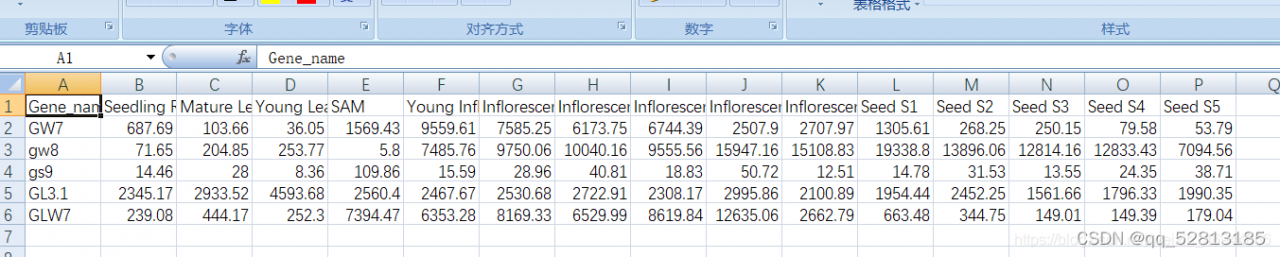
1.数据的获取–从NCBI数据库下载基因表达谱数据
2.数据整理–将所有下载的基因表达谱数据放在一个Excel里,如下
再转成文本文档(grain2.txt)。
3.R语言绘制热图(直接复制>后的代码)
getwd() 查看当前工作目录 setwd(“D:/1-R/myfile/file4”)将工作目录设为grain2文本所在文件夹
install.packages(‘pheatmap’)
data<-read.table(“grain2.txt”,head=T)
attach=data
row.names(data)<-data$Gene_name
data<-data[,-1]
data<-data.matrix(data)
library(pheatmap)
pdf(“2222.pdf”,height=60,width=60) ``输出的图PDF存于所设定的文件夹
pheatmap(data,cluster_cols=FALSE,clustering_distance_row=“correlation”,clustering_method=“complete”,color=colorRampPalette(c(“green”,“black”,“red”))(100),revC=FALSE,scale=“row”,margins=c(5,10),fontsize_row=8,cellheight=30,cellwidth=50)
dev.off()
结果显示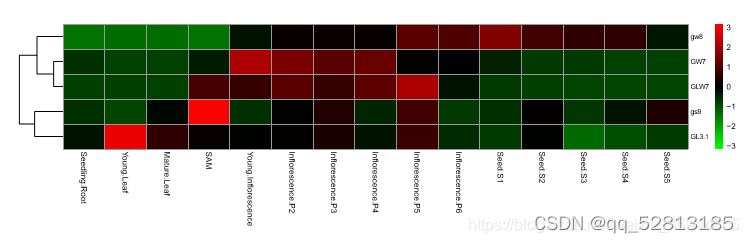
版权声明:本文为qq_52813185原创文章,遵循CC 4.0 BY-SA版权协议,转载请附上原文出处链接和本声明。